
La Unidad de Bioinformática, Bioestadística y Biología Computacional de la RAI ofrece a la comunidad científica estudios in-silico, orientados hacia el análisis e interpretación de resultados experimentales sobre sistemas de interés biomédico. Estos estudios son obtenidos por diversas técnicas como análisis bioquímicos, acoplamiento molecular (BIACORE), secuenciación masiva de nueva generación y mutagénesis dirigida, entre otras.
La especialidad del grupo de trabajo en la Unidad incluye el análisis de datos de secuenciación masiva para el el ensamble de genomas guiados por referencia y de novo, el análisis de transcriptomas, proteomas y metagenomas, así como la identificación y anotación de genes y/o dominios, la visualizacion tridimensional de biomoléculas, el modelado de proteínas, el análisis de interacciones proteína-ligando, el análisis de trayectorias de dinámica molecular y la aplicación e interpretación de múltiples técnicas estadísticas a los datos. La Unidad también se encarga del diseño de experimentos y muestreo, del análisis exploratorios de los datos y de la aplicación de nuevas metodologías en bioestadística.
En resumen, integramos la bioinformática, la bioestadística y la biología computacional como una parte fundamental en los protocolos de investigación dentro del campo de la biomedicina, a fin de fortalecer la investigación multidisciplinaria en el estudio del funcionamiento de los sistemas vivos.
Fundamentos
Bioinformática
La secuenciación masiva de última generación produce una gran cantidad de datos al final de cada experimento. Una sola corrida puede generar imagenes digitales que llegan a requerir hasta 3 TB de espacio en disco, las cuales se procesan para obtener archivos de secuencias de aproximadamente 100 GB. Por lo tanto, la infraestructura computacional en las técnicas de secuenciación masiva es una parte esencial para la recolección, almacenamiento y evaluación de calidad de los datos. El manejo e interpretación de información digital generada en los secuenciadores requiere de personal especializado en herramientas de cómputo de alto rendimiento, elaboración de scripts de análisis bioinformático y bioestadístico, uso y generación de bases de datos, entre otras herramientas.
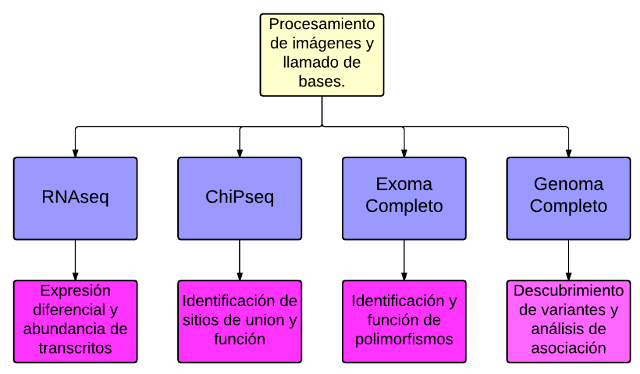
El procesamiento de datos incluye tres niveles:
Análisis primario. Consiste en el procesamiento de imágenes por medio del software del fabricante, el llamado de bases y el control de calidad de las secuencias.
Análisis secundario. Consiste en el alineamiento y amapeo de las secuencias para la identificación y posterior ensamblado de genomas y/o transcriptomas.
Análisis terciario. Este tipo de análisis es más especializado e incluye particularidades de cada tipo de experimento. Consiste en identificar variantes, análisis estadísticos de asociación, análisis de correlaciones relevantes para la función biológica, etc.
El siguiente diagrama muestra las etapas del procesamiento de datos que se lleva a cabo en la Unidad de Bioinformática, Bioestadística y Biología Computacional.
Bioestadística
Las conclusiones, decisiones y acciones en una investigación son dirigidas por los datos, de la misma forma en que el objetivo de la investigación dirige el método y las estrategias a seguir para la obtención de los datos. Por tal razón, nos dedicamos a la aplicación más adecuada de los métodos estadísticos tanto para el diseño del muestreo como para el análisis de los resultados. El análisis de los datos nos permite describir e interpretar fenómenos biológicos así como inferir sus posibles relaciones causales.
La bioestadística incluye diversas disciplinas y niveles de organización en los sistemas. Particularmente, la medicina y la biomedicina representan un campo muy extenso para la aplicación de los métodos estadísticos y una oportunidad de investigación conjunta.
Biología computacional
Por los importantes avances de los últimos años, tanto en métodos numéricos como en la capacidad de cómputo, estamos frente a la nueva era tecnológica que está revolucionando nuestra comprensión de los procesos celulares a nivel molecular. A partir de la generación de trayectorias de simulación para sistemas de alrededor de 100 mil átomos y durante ~1 microsegundo, es viable describir las funciones de biomoléculas como proteínas, ácidos nucleicos, lípidos, azúcares o fármacos. Las aplicaciones potenciales de este conocimiento incluyen nuevas propuestas de estrategias terapéuticas para enfermedades crónicas y/o degenerativas como el cáncer, el síndrome metabólico, la diabetes mellitus y la enfermedad de Alzheimer, entre otras.
Equipos
La unidad cuenta con:
Acceso a supercomputo UNAM (Cluster). Un servidor con 64 núcleos, 512 Gbytes de RAM y 8.5 TB de almacenamiento en disco duro. Unidad de almacenamiento de 18 TB (Intel Xeon E5-2600). Estaciones de trabajo (Workstation Z230 i7) para uso de los miembros de la RAI.
| Manual de procedimientos de los servicios
|
Servidor de cómputo |
| Manual de procedimientos
|
Unidad de almacenamiento |
Contacto
Grupo de trabajo
Para mayor información o asesoría sobre los servicios favor de contactar a:
-
Dra. Nancy R. Mejía Domínguez
Investigador Responsable (Bioestadística y Bioinformática)
- nmejiacic.unam.mx
Página personal: Biostatistics RAI-UNAM
-
Dra. Georgina Hernández Montes
Técnico Académico (Bioinformática)
- yinnacic.unam.mx
-
M. en C. Tobías Portillo Bobadilla
Técnico Académico (Bioinformática)
- tobiascic.unam.mx
- +01 (55) 5487 0900 Ext. 6327
Biología de sistemas:
-
Dr. Osbaldo Resendis-Antonio
Profesor e Investigador
- resendiscic.unam.mx
Laboratorio de Biología de Sistemas para Enfermedades Humanas (INMEGEN)
Página personal: resendislab.github.io
